Visualize my spectra
光谱系列第1辑: 光谱可视化
从去年开始就一直在分析光谱的数据,刚开始以为R里面会有包能解决我关于光谱的所有问题。经过一段时间的试错之后,我发现自己错了。
于是记录下来我自己实践的过程。
批量读取光谱数据
一般来说,拿到的原始光谱数据数据有这样的特点:
- 光谱反射率和光谱吸收值夹杂在文件内部。 以SR3501的数据为例,打开之后是这样的(如下图所示)。文件内部有很多关于仪器和配置的信息。真正对我们有价值的数据在27行以后。具体的就是此处的两列数据,第一列为波长,第二列为对应的反射率。所以这就要求读取数据的时候找到指定的行。

2.数据文件不止一个,常常需要批量读取。
于是经过多次试错与总结,写下了下面的方法批量读取指定行的光谱数据。
# get file names of all spectra
dir = "Spec/"
files = list.files(path = dir ,recursive = TRUE)
# read spectra from AB files
filepath = paste(dir, files, sep = '')
# define a dataframe to store spectra data
spec.bdf <- data.frame()
for (flp in filepath) {
spec <- read.table(flp, skip = 27, header = F ,
stringsAsFactors = F,row.names = 1)
spec.t <- as.data.frame(t(spec))
spec.bdf <- rbind(spec.bdf, spec.t)
}
根据需求,有时候要对数据做一下转置。转置后再把所有的数据bind到一起。
采用ggplot2绘图,需要的是长数据,原来两列的数据正好符合要求,只需要添加一列信息即可。
# get file names of all spectra
dir = "Spec/"
files = list.files(path = dir ,recursive = TRUE)
# read spectra from AB files
filepath = paste(dir, files, sep = '')
# define a dataframe to store spectra data
spec.bdf <- data.frame()
for (flp in filepath) {
spec <- read.table(flp, skip = 27, header = F ,stringsAsFactors = F)
colnames(spec) <- c("Wavelengths","Reflectance")
spec$unique_id <- basename(flp)
spec.df <- rbind(spec.df, spec)
}
有了数据就可以展示了,下面是选出来100条光谱进行的展示。
library(ggplot2)
library(cowplot)
load(file = "D:/blogdown/sourceRda/show spectra.rda")
# Define nice breaks for x axis
brk <- pretty(as.numeric(unique(spec$Wavelengths)), n = 10)
spec.df$grp <- "A"
ggplot(spec.df, aes(Wavelengths, Reflectance,group = unique_id)) +
geom_line(aes(color = grp),alpha = 0.7, size = 0.6) +
scale_x_continuous(breaks = brk) +
theme_cowplot() +
scale_color_manual(values = "black") +
theme(legend.position = "none") # remove the legend
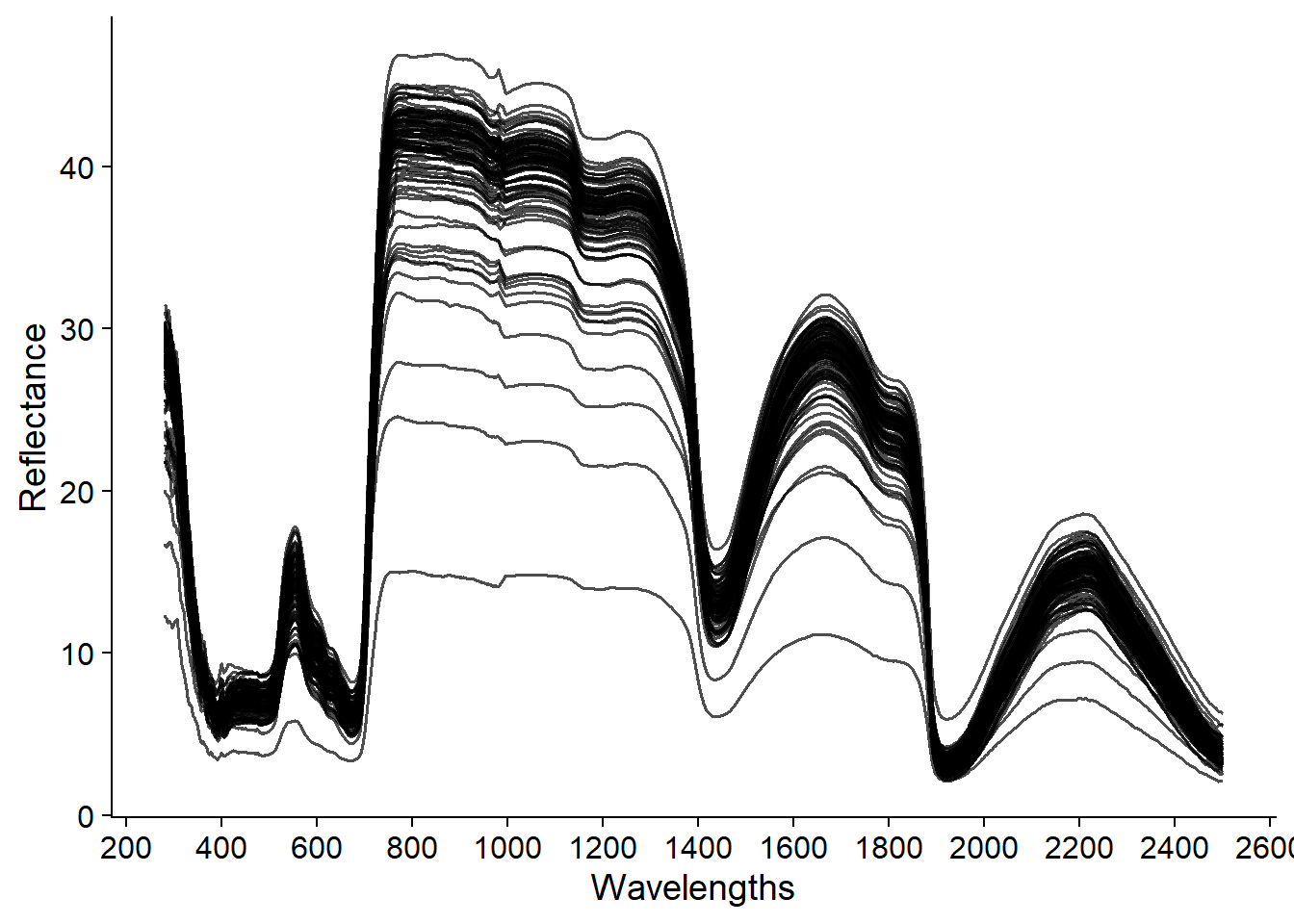
为了好看,我们还可以给每条光谱加上颜色
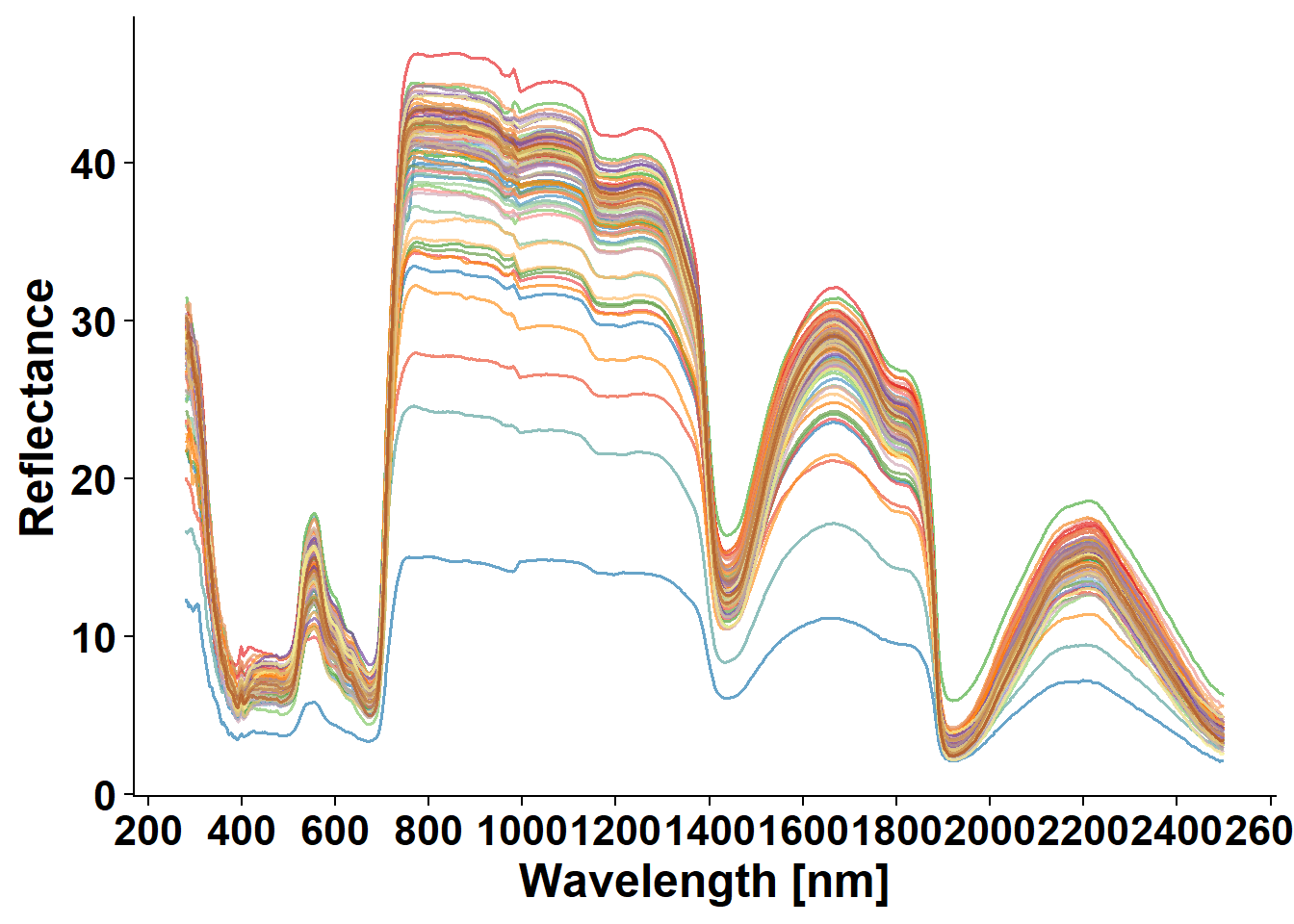
后续更多有意思的分析,且听下回分解
先附上几张图,再留着以后慢慢讲
- 如何优雅的展示不同类别的光谱

- 光谱定量分析与主成分分析

- 主成分分析用于分组分类
